Eugene Douglass, Practically Science
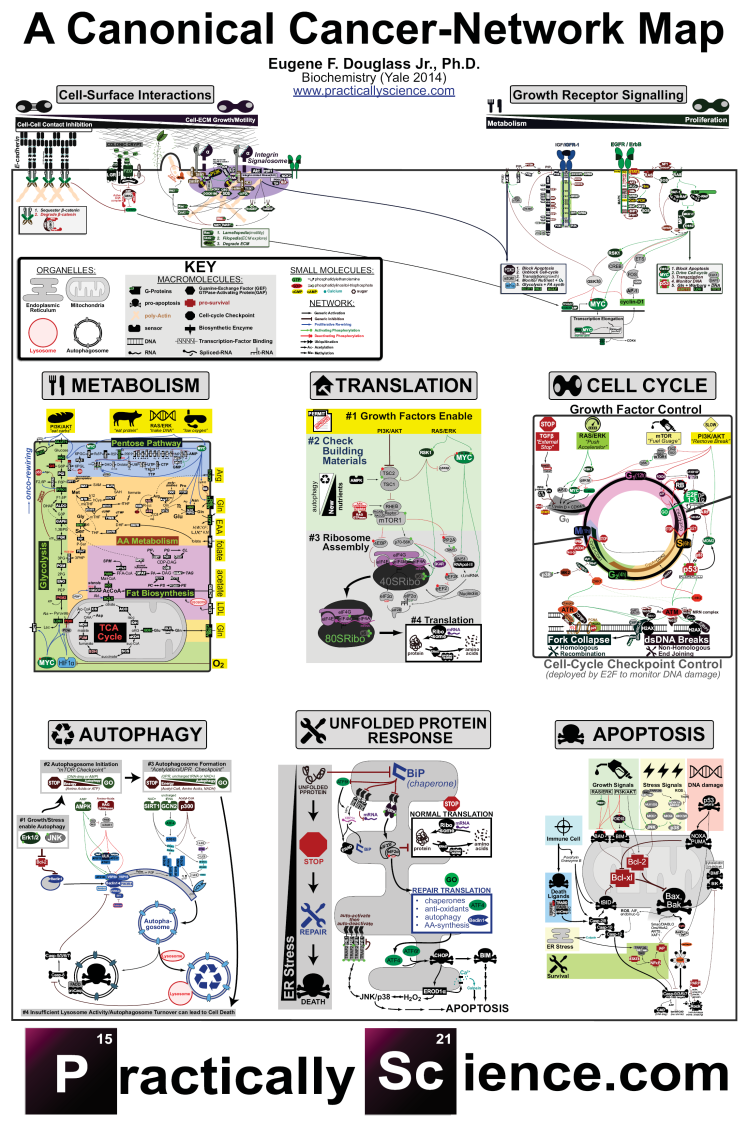
Kanser, normal hücresel devrelerin mutasyon veya epigenetik olarak değiştirilerek yeniden programlamasını yansıtan en az 10 farklı “Belirteç” tarafından tanımlanan karmaşık bir hastalıktır.
HÜCRE YÜZEYİ ETKİLEŞİMİ: Bu modül, diğer hücrelerin ve hücre dışı matrisin (ECM) bir kanser hücresinin çoğalma ve çoğalma kararlarında sahip olduğu rekabetçi etkileri vurgulamak için hazırlanmıştır. Genelde, E-kadherin aracılığıyla Hücre-Hücre Bağlanması anti-proliferatiftir; Buna karşılık, Cell-ECM Entegrasyon yoluyla olan etkileşimler, “Integrin Sinyalozomu” olarak adlandırdığım çok bileşenli bir kompleks aracılığıyla büyüme hormonu benzeri bir sinyal yoluyla proliferatif olabilir.
BÜYÜME RESEPTÖR SİNYAL YOLAĞI: Bu modül, farklı büyüme hormonu reseptörlerinin İnsülin benzeri Büyüme Faktörü Reseptörü (IGFR) ve Epidermal Büyüme Faktörü Reseptörü (EGFR) arasındaki farklı sinyallemeyi vurgulanması için çizilmiştir. Her iki reseptör Fosfoinositid 3-kinaz (PI3K) ve RAS / RAF / MEK Yollakları vasıtasıyla sinyal yeteneğine sahipken, indükleme kapasitelerinde değişiklik gösterirler. Örneğin, RAS / RAF / MEK sinyal yolağında daha etkili olan EGFR ile karşılaştırıldığında, IGFR bir iskele proteini olan IRS1 vasıtasıyla PI3K sinyal yolağında daha etkili olur.
METABOLİZMA: Bu modül, metabolik enzimatik “montaj hatlarını” 5 alt modül halinde bölerek anlatılmıştır:
- Glikoliz (enerji ve hücre yapı taşları i için şekerlerin parçalanması)
- Pentoz Fosfat Yolağı (DNA ve RNA’nın yapısına giren şekerlerin ve redoks homeostazını korumak için şeker ve amino asitlerin kullanılması)
- Amino Asit (AA) Metabolizması (AA’lerin enerji için parçalanması ve biyosentezi)
- Yağ Biyosentezi (sterollerin, sfingomyelin (SPM), Fosfatidiletanolamin (PE), Fosfatidilkolinin (PC), Fosfatidilserin (PS), Triasilgliserol (TAG), Fosfatidilinozitol fosfatların (PIPx) biyosentezi
- TCA Döngüsü (maksimum enerji eldesi için şeker ve amino asitlerin kullanılması).
Bunlara ek olarak, metabolizmanın yönünü değiştiren sinyal inputları üstte sarı renkle vurgulanırken, besinsel inputlar sağ tarafta sarı ile vurgulanmıştır (EAA: Esansiyel Amino Asitler; LDL: düşük yoğunluklu lipoprotein). Kanserde aşağı regüle edilenler kırmızı başlıklar (örn., TCA döngüsü), kanserde yukarı doğru düzenlenen genler ve yollaklar yeşil başlıklarla belirtilmişlerdir (örn., Glikoliz, Pentoz Fosfat Yolağı, AA Metabolizması, Yağ Biyosentezi).
TRANSLASYON: Bu modül, bir binaya benzetilerek yapısal malzemelerden binanın yapılmasının başlatılmasını açıklamaktadır: İlk olarak, Büyüme Faktörleri, hücrenin “yapı izni” ni onaylar. İkincisi, mTOR, “yapı malzemeleri” nin (örneğin, besin maddeleri) yeterli olup olmadığını denetler. Değilse eski malzemeler otofaj yoluyla geri dönüştürülür. Üçüncü olarak, ribozom (“iş makinaları”) monte edilir. Dördüncüsü, “inşaat” başlar.
HÜCRE DÖNGÜSÜ: Bu modül, 4 aşamaya bölünen hücre döngüsünün döngüsel yapısını vurgulamak için çizilmiştir: G1 (Büyüme fazı 1 yaklaşık 12 saat), S (Sentez yaklaşık 6 saat), G2 (Büyüme fazı 2 yaklaşık 4 saat), M ( Mitoz yaklaşık 1 saat). Döngünün üst yarısı büyüme faktörleri tarafından kontrol edilirken, alt yarısı ise hücre bölünmesi sırasında oluşan DNA hasarını “hücre döngüsü kontrol noktası mekanizması” ile kontrol eder.
- TGFβ yolağı “firen” işlevi görür. Bir hücre topluluğu bu yolaktan aldığı sinyale bağlı olarak büyümesini durdurur.
-
RAS / RAF / MEK / ERK yolağı “gaz pedali” işlevi görür. Bu yolak hücre döngüsünü başlatan siklin-D ile koreledir.
-
PI3K / AKT yolağı yakıt göstergesi olan mTOR’u devreye sokar ve kontrol noktalarındaki firenler olan ve MYC-sinyali bağlamında çoğalmayı önleyen FOXO ve p53 transkripsiyon faktörlerini ortadan kaldırır.
Son olarak, Hücre Döngüsü Kontrol Noktaları, çoğunlukla, çift sarmallı DNA (dsDNA) kırıkları ve DNA replikasyon çatalı bozulmaları etrafında oluşan çok bileşenli kompleksler tarafından indüklenir. Çifte zincir DNA kırıkları Homolog Olmayan Uç Bağlamaları veya Homolog Rekombinasyon ile onarılabilirken, replikasyon çatalı bozulmaları Homolog Rekombinasyon ile onarılır.
OTOFAJİ: Bu modül, otofajinin kademeli olarak işlemesini vurgulamak için çizilmiştir:
- Büyüme veya Stres sinyalleri otofajiyi etkinleştirir.
- Ortam besin maddleri bakımından fakirse mTOR otofajiyi başlatır.
- Hücrenin enerji durumu (Acetyl-CoA, NAD +) veya protein stresi (Yanlış Katlanmış Proteinlere Cevap olarak), işlevsel olmayan sitoplazmik kompartmanların çevresinde otofagozomların oluşumunu kolaylaştırır.
- Lizozomal aktivite ile işlevsel olmayan sitoplazmik materyal geri dönüştürülür.
YANLIŞ KATLANMIŞ PROTEİN YANITI: Bu modül, Endoplazmik Retekulum (ER)’da yanlış katlanmış proteinlere hücresel bir cevap olarak ortaya çıkan mekanizmayı betimlemektedir.
- ER’de olan protein translasyonu (protein sentezi) durdurulur.
- Modifiye Onarım translasyonu başlar.
- Onarım başarısız olursa apoptoz başlatılır.
APOPTOZ: Hücrenin kendini intiharı, yani apoptoz; büyüme faktörleri, stres, DNA hasarı ve bağışıklık sistemi de dahil olmak üzere çeşitli sinyallerden etkilenen karmaşık bir süreçtir. Genel olarak, mitokondriyal içerikler olan Bax ve Bak proteinleri “cehennemin giriş “kapılarıdır” ve bir hücrede ölüme yol açarlar. BCL-xl ve BCL-2 gibi anti apoptotik proteinler ise “o kapıda kilit” olarak düşünülebilirler.
- Büyüme Faktörü Sinyalleri, hücrenin korunması için kapıyı güçlendiren ekstra “cıvatalar” olarak düşünülebilir
- DNA hasarı ve stres ise kapıyı açıp hücrenin ölümüne sebep olan “anahtarlar” olarak düşünülebilir
- Bağışıklık hücreleri ve Ölüm Ajanları (Ligandlar) (örn., TNF, FASL) kendi “anahtarları” na sahiptir, ancak bunlar doğrudan kaspaz aktivasyonu yoluyla “arka kapıdan” da girebilirler.
REFERANSLAR
- Weinberg, R.A. The Biology of Cancer (2nd ed.), 2014, Garland Science.
- Murphy, K. Janeway’s Immunobiology 8th Ed. Garland Science 2012
- Alberts, B. Molecular Biology of the Cell 5th Ed. Garland Science 2008
- Goldberg, S. Clinical Biochemistry made ridiculously simple Medmaster Inc. 1997
- Milo, R.; Phillips, R. Cell Biology by the Numbers Garland Science 2015
- Devita, V.T.; Lawrence, T.S.; Rosenberg, S.A. Cancer: Principles and Practice of Oncology 10th Ed. Wolters Kluwer. 2015
- Berridge, M.J. Cell Cycle and Proliferation Cell Signalling Biology 2014; doi:10.1042/csb0001009
- Kang, R.; Zeh, H.J.; Lotze, M.T.; Tang, D. The Beclin 1 network regulates autophagy and apoptosis. Cell Death and Differentiation, 2011, 18, 571.
- Rubinstein, A.D.; Kimchi, A. Life in the balance – a mechanistic view of the crosstalk between autophagy and apoptosis J. Cell Sci., 2012 125, 5259.
- Galluzzi, L.; Pietrocola, F.; Levine, B.; Kroemer, G. Metabolic Control of Autophagy Cell, 2014, 159, 1263.
- Green, D.R.; Levine, B. To Be or Not to Be? How Selective Autophagy and Cell Death Govern Cell Fate. Cell, 2014, 65.
- Ouyang, L.; Shi, Z.; Zhao, S.; Wang, F.-T.; Zhou, T.T.; Liu, B.; Bao, J.-K. Programmed cell death pathways in cancer: a review of apoptosis, autophagy and programmed necrosis Cell Prolif., 2012, 45, 487.
- Reinhardt, H.C.; Schumacher, B. The p53 network: cellular and systemic DNA damage responses in aging and cancer Trends in Genetics, 2012, 28, 128.
- Cao, S.S.; Kaufman, R.J. Primer: Unfolded Protein Response. Current Biology, 2012, 22, R622.
- Hetz, C. The unfolded protein response: controlling cell fate decisions under ER stress and beyond Nat. Rev. Mol. Cell Bio., 2012, 13, 89.
- Walter, P.; Ron, D. The Unfolded Protein Response: From Stress Pathway to Homeostatic Regulation Science, 2011, 334, 1081.
- Cantor, J.R.; Sabatini, D.M. Cancer Cell Metabolism: One Hallmark, Many Faces Cancer Discovery, 2012, OF1.
- Yizhak, K.; Chaneton, B.; Gottlieb, E.; Ruppin, E. Modeling cancer metabolism on a genome scale Mol. Sys. Bio., 2015, 11, 817.
- Pavlova, N.N.; Thompson, C.B. The Emerging Hallmarks of Cancer Metabolism Cell Metab., 2016, 23, 27.
- Cairns, R. A.; Harris, I.S.; Mak, T.W. Regulation of cancer cell metabolism Nat. Rev. Cancer, 2011, 11, 85.
- Shadel, G.S.; Horvath, T.L. Mitochondrial ROS Signaling in Organismal Homeostasis Cell, 2015, 163, 560.
- Jones, D.P. Radical-free biology of oxidative stress Am. J. Physiol. Cell Physiol., 2008, 295, C849.
- Hanahan, D.; Weinberg, R.A. Hallmarks of Cancer: The Next Generation Cell, 2011, 144, 646.
- Kolch, W.; Halasz, M.; Granovskaya, M.; Kholodenko, B.N. The dynamic control of signal transduction networks in cancer cells Nat. Rev. Cancer, 2015, 15, 515.
- Cully, M.; Downward, J. Snapshots: Ras Signalling Cell, 2008, 133, 1292.
- Frisch, R.; Downward, J. Snapshots: Class I PI3K Isoform Signalling Cell, 2013, 154, 940.
- Carracedo, A.; Salmena, L., Pandolfi, P.P Snapshots: PTEN Signaling Pathways Cell, 2008, 133, 550.
- MacDonald, B.T., Semenov, M.V., He, X. Snapshots: Wnt/beta-catenin Signaling Cell, 2007, 131, 1204.
- Semenov, M.V., Habas, R.; MacDonald, B.T., He, X. Snapshots: Wnt/beta-catenin Signaling Cell, 2007, 131, 1378.
- Kadowaki, T.; Ueki, K.; Yamauchi, T.; Kubota, N. Snapshots: Insulin Signaling Pathways Cell, 2012, 148, 624.
- Trempolec, N.; Dave-Coll, N.; Nebreda, A.R. Snapshots: p38 MAPK Signaling Cell, 2013, 152, 656.
- Jin, M.; Liu, X.; Klionsky, D.J. Snapshots: Selective Autophagy Cell, 2013, 152, 368.
- Wiseman, R.L. Haynes, C.M.; Ron, D. Snapshots: The Unfolded Protein Response Cell, 2010, 140, 590.
- Spizzo, R.; Nicoloso, M.S.; Croce, C.M.; Calin, G. Snapshots: MicroRNAs in Cancer Cell, 2009, 137, 586.
- Xenia, M.; Iiagan, G.; Kopan, R. Snapshots: Notch Signaling Pathway Cell, 2007, 128, 1246.
- Crabtree, G.R.; Schreiber, S.L. Snapshots: Ca2+-Calcineurin-NFAT Signaling Cell, 2009, 138, 210.
- Morgan, D.O.. Cell Cycle Regulators I Cell, 2008, 135, 764.
- Morgan, D.O.. Cell Cycle Regulators II Cell, 2008, 135, 974.
- Pope, B.D.; Aparicio, O.M.; Gilbert, D.M. Snapshots: Replication Timing Cell, 2013, 152, 1390.
- Verbrugge, I.; Johnstone, R.W.; Smyth, M.J. Snapshots: Extrinsic Apoptosis Pathways Cell, 2010, 143, 1192.
- Salvesen, G.S.; Ashkenazi, A. Snapshots: Caspases Cell, 2011, 147, 476.
- Kruse, J.-P.; Gu, W. Snapshots: p55 Post Translational Modifications Cell, 2008, 133, 930.
- Hardwick, J.M. Youle, R.J. Snapshots: BCL-2 Proteins Cell, 2009, 138, 404.
- Soulard, A.; Hall, M.H. Cell Snapshots: mTOR Signalling Cell, 2007, 129, 434.
- Laplante, M.; Sabatini, D.M. mTOR Signalling at a Glance J. Cell Sci. , 2009, 122, 3589.
- Reed, J.C.; Huang, Z. Apoptosis Pathways and Drug Targets Nature Reviews Drug Discovery/Molecular Cell Biology , 2004, http://www.nature.com/reviews/poster/apoptosis
- Conacci-Sorrell, M.; Zhurinsky, J.; BenZe’ev, A. The cadherin-catenin adhesion system in signaling and cancer, J. Clin. Invest., 2002, 109, 987.
- Cheng, Z.; Tseng, Y.; White, M.F. Insulin signaling meets mitochondria in metabolism Trends in Endocrinology and Metabolism, 2010, 21, 589.
- Hanke, S.; Mann, M. The Phosphotyrosine Interactome of the Insulin Receptor Family and Its Substrates IRS-1 and IRS-2* Mol Cell. Proteomics, 2008, 8.3, 519
- Harburger, D.S.; Calderwood, D.A. Integrin Signalling at a Glance J. Cell Sci., 2009, 122, 159.
Kanser 101: temel öğeler
Reblogged this on tabletkitabesi.
BeğenBeğen